我国学者在拓扑编程DNA折纸领域取得进展
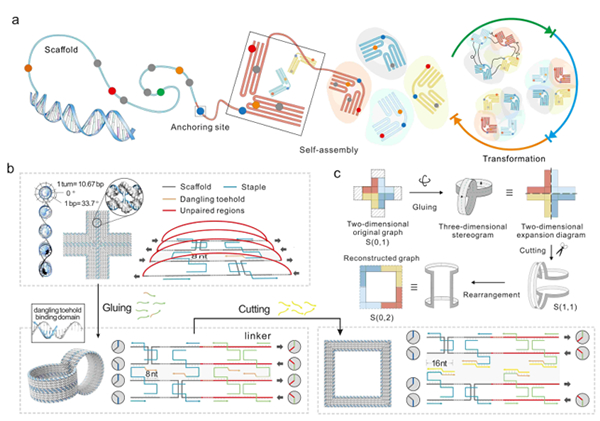
图 基于DNA折纸系统的拓扑转换与计算
在国家自然科学基金项目(批准号:T2188102、21991134)等资助下,上海交通大学樊春海教授与华东师范大学裴昊教授合作开发了一种拓扑编程的DNA折纸系统,这种拓扑变构的DNA折纸可形成动态支架,在纳米尺度上编码信号节点的连接模式与连通性,建立拓扑图形计算新模式。研究成果以“在拓扑编程DNA折纸上编码信号传播(Encoding signal propagation on topology-programmed DNA origami)”为题,于2024年6月17日发表在《自然•化学》(Nature Chemistry)杂志,论文链接:https://www.nature.com/articles/s41557-024-01565-2。
如何利用拓扑变换来动态配置节点之间的连接,建立适应性计算功能连接的拓扑模式,是图形计算的前沿方向。近年来,结构DNA纳米技术的发展为构建仿生拓扑异构纳米结构提供了新机遇。然而,如何构建具有复杂拓扑调控功能并能实现复杂拓扑变换的人工结构仍然是一个巨大的挑战。
针对这一挑战,研究团队首先提出了一种在分子层面实现拓扑操作(粘贴和剪切)的策略,可促使DNA折纸结构发生全局构象变化。基于该策略成功设计构建了三种类型的可重构DNA折纸系统,通过分子拓扑操作实现了结构的连续拓扑变化,并通过拓扑不变量(包括亏格、边界数目和可定向性)表征了这些拓扑变化。这些拓扑编程的DNA折纸可作为动态支架用于时空可控的分子信号传播,拓扑转换可改变节点的位置、连接模式与连通性,建立拓扑性质与计算网络架构的直接关联,实现拓扑性质变化调节计算功能,使网络拓扑适应功能连接实现多种逻辑计算。
基于分子拓扑连续变换的机制,本研究为探索纳米尺度上的拓扑图形化计算提供了可编程动态支架,实现了DNA折纸结构的连续拓扑变换,并在实验上展示了高达77个分子节点的信号传递网络规模。基于拓扑变构实现计算网络节点连接模式的变换,在纳米尺度展现出核酸分子极高的拓扑可编程性,取得了DNA纳米技术领域的新突破。通过多达100多个链置换反应和杂交反应在分子水平上实现了高效的拓扑操作。利用空间上排列活性DNA发夹,实现了信号在3D折纸曲面上不同长度(超过300 nm)、方向和曲率(凹或凸)的传输路径上的传播。本研究提出的拓扑操作策略为制造动态DNA自组装提供一条通用途径,并为推动拓扑图形化计算提供了新思路。


 电话:400-9933-062
电话:400-9933-062 电子邮箱:business@wykt.com
电子邮箱:business@wykt.com

